基因剔除,基因體編輯
基因剔除動物的遺傳分析
基因剔除動物是指編碼目標基因的序列經過人為刪除的動物,分析目的在於比較這類動物與正常群體的遺傳功能,確立基因缺失引發疾病的動物模型。
使用基因體編輯工具確認基因剔除

CRISPR/CAS 及 TALEN 等基因體編輯工具推出後,可以更輕鬆刪除和插入序列,比傳統方法簡單許多。不過,以 DNA 定序進行編輯後確認,一向都有費時且成本高昂的問題。
以下舉例說明一個極其簡單的實驗流程,使用 DNA/RNA 分析專用 MCE-202 MultiNA 微晶片電泳系統確認目標基因是否發生缺失或插入。
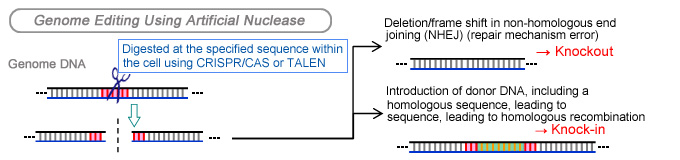
分析原理/方法
完成基因體編輯後,以 PCR 擴增含有缺失點 (deleted point) 的區域。如下所示,將 PCR 產物變性處理,再黏合形成異源雙股體 (heteroduplex)。此結構變化會造成泳動模式不同,之後以 MultiNA 量測,確認是否存在缺失 (deletion)。此方法還有一項額外的優點,能夠辨識較短的缺失 (deletion),這類缺失難以從片段的長度差異來確定。
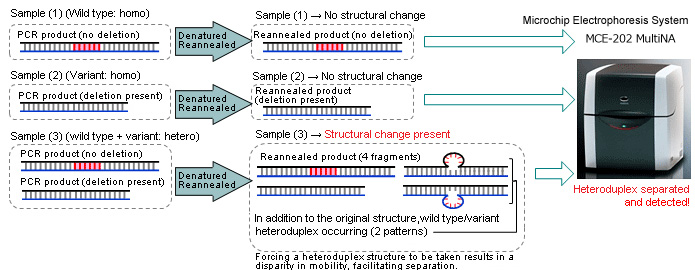
分析範例
鑑定以 TALEN 編輯的同型/異型變體
將 Medaka Oryzias laptipes (異型變體)與經TALEN 編輯後的變體 (刪除 8 bp)雜交,對雜交群體的缺失區域進行 PCR,再使用 MultiNA 分析異源雙股移動率。


分析方法/數據提供:京都大學農業部 Masato Kinoshita 博士
Citation: Ansai, S. et al., Biol Open. 2014 Apr 11;3(5):362-71.(CRISPR)
Citation: Ansai, S. et al., Dev Growth Differ. 2014 Jan;56(1):98-107.(TALEN)
使用 Ampdirect 進行大鼠血液基因分型
Ampdirect 可抵消蛋白質及醣類等 PCR 抑制劑,便於讓樣品 (包括原始狀態) 直接進行 PCR, 無需純化模板 DNA。以下我們舉例說明使用 Ampdirect 和 MultiNA 對 FTA ® 卡上的大鼠血液進行快速基因分型。
分析範例
SERs (自發性癲癇大鼠) 動物模型用於探討人類的癲癇,包括嚴重的失神性癲癇發作及強直型痙攣等症狀。京都大學醫學院實驗動物中心以雜交 tremor rat 和 zitter rat兩個突變品系的基因,開發出 SER,而其中的 tremor ( tm) 與 zitter ( zi) 突變皆為同合子 (homozygous)。tm 突變是 Aspa (天冬胺酸醯化酶 aspartoacylase) 基因中的基因體缺失 (大約 200 kbp),而 zi 是 Atrn (attractin) 基因中的8 bp 缺失。以 zi 同合子 (homozygous) 及 tm 異合子 (heterozygote) 的手足交配,維持此品系。 這些缺陷必須被檢測出來,才能夠選擇適當的親代大鼠以產生子代。在這兩個突變基因中,tm 突變呈現約 200 kbp 的較長缺失,我們使用兩對引子進行多重 PCR,以檢測是否存在這項缺陷。 一對引子用於野生型檢測,使用黏合於這個缺失區域的 (deletion region) 引子進行擴增,如果不存在缺失 (deletion) 才能取得 PCR 擴增產物。tm 基因缺失檢測的另一對引子,則是黏合於出現缺失的區域,如果存在缺失時才能繼續取得 PCR 擴增產物 (約 240 bp)。由於此區域非常長 (約 200 kbp),如果不存在缺失,就無法產生 PCR 擴增產物。
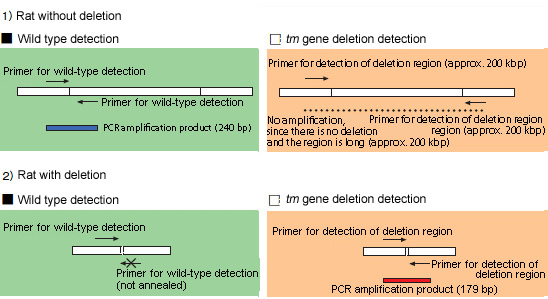
分析結果彙整說明於下表。

結果
MultiNA 分析 (使用的試劑組:DNA-500 預混合模式)
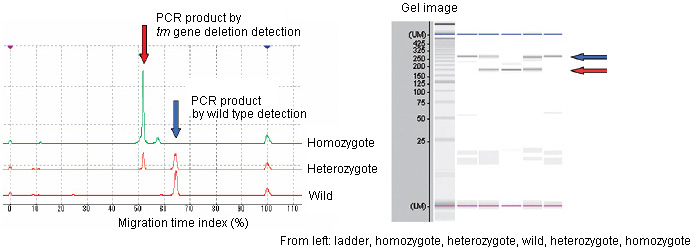
|
樣品提供: |
京都大學醫學院實驗動物中心 Takashi Kuramoto 副教授 /p> |
Microchip Electrophoresis System for DNA/RNA

微晶片電泳系統可有效地快速地分析 DNA 和 RNA 核酸樣品大小並定量。與瓊脂糖凝膠電泳相比,可重複使用的高性能微晶片的分析成本更低。只需置入樣品和分離緩衝液即可執行多達 120 次無人自動分析。
Ampdirect Plus DNA Amplification Reagent

血液及動物組織含有大量抑制酵素反應的物質,因此需從樣品純化出 DNA,再以 PCR 進行 DNA 分析。
Ampdirect Plus DNA 擴增試劑 (PCR 緩衝液) 可限制 PCR 抑制物質在樣品中的作用,無需純化 DNA 即可對血液、動物/植物組織、SDS 等樣品進行 PCR。


