確認基因體編輯產生的突變

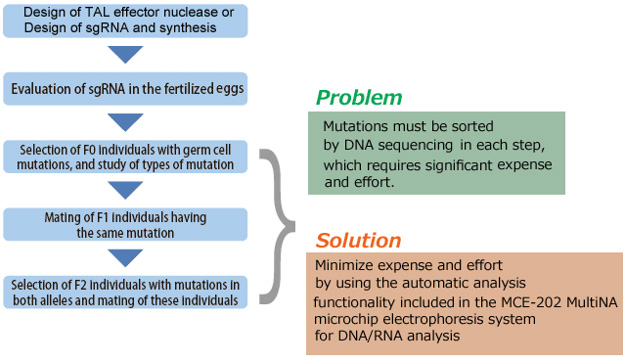
使用基因體編輯以建立突變體,需要在各種程序對突變進行分類,不過確認突變的技術在成本和操作效率方面,仍有一些問題。本文說明一種有效確認突變的程序,基於異源雙股泳動試驗 (Heteroduplex Mobility Assay, HMA),使用 Shimadzu MCE-202 MultiNA 微晶片電泳系統進行 DNA/RNA 分析。
基因體編輯
基因體編輯是指使用人工核酸酶的基因改造技術 (用於破壞基因體特定位置的核酸),以刪除、插入或替換部分基因體序列,產生所需的突變。目前,zinc-finger 核酸酶 (zinc-finger nucleases, ZFN)、類轉錄活化因子核酸酶 (transcription activator-like effector nucleases, TAL 核酸酶)、叢集規律間隔短迴文重複序列相關蛋白 9 (clustered regularly interspaced short palindromic repeats associated protein 9, CRISPR/Cas9),是做為基因體編輯工具的主要人工核酸酶。其中,第三代 CRISPR/Cas9 核酸酶由於容易設計目標序列,因此迅速普及。
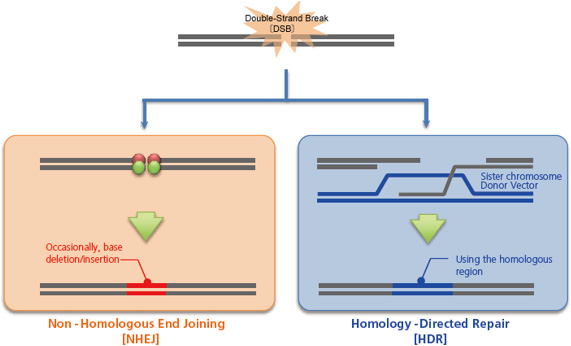
這些人工核酸酶或表現載體,以顯微注射、電穿孔或其他技術注入細胞後,會插入目標序列處的基因體。隨後細胞會進行修復,主要有兩種修復途徑,一是與細胞週期無關的非同源末端接合 (non-homologous end joining, NHEJ),此路徑直接將斷裂的基因體接合,暫時修補基因體斷裂的問題。不過,過程中可能造成一些鹼基的缺失或插入,這種隨機生成的突變,可用於刻意誘發框移 (frameshift) 或基因破壞 「剔除」(knockout)。
另一個路徑是同源定向修復 (homology-directed repair, HDR),以正常的姐妹染色體為模板引導修復,此路徑可將基因體修復至原有狀態,不會造成非同源末端接合那樣的突變類型。然而,以同源雙股 DNA (或單股 DNA) 為供體而注入細胞,基因體結合是來自供體的序列,進而達到基因插入「敲入」(knock-in) 或基因修飾的結果。
基因體編輯的基因改造方法,相較於過去依賴自然隨機重組的方法,大幅提高了修飾效率。此外,
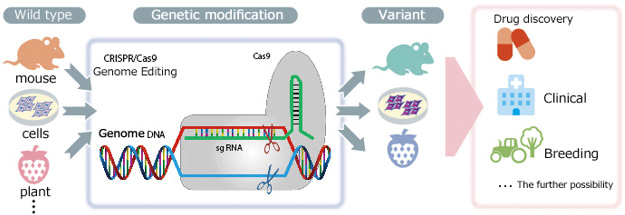
過去只能對小鼠及其他可使用胚胎幹細胞的動物進行基因改造,現在利用基因體編輯,其他動物的基因也能夠改造。在這之前,對其他動物進行改造一直是巨大的挑戰,例如魚類、兩棲動物、爬蟲類、大型哺乳動物等,如今基因體編輯甚至可用於昆蟲和植物的基因改造。世人非常期待基因體編輯不僅停留在基礎研究層面,除了應用在醫學領域 (例如建立人類基因體疾病模型或基因療法),更能擴大至改良植物和動物品種。
建立突變及突變體的流程
本節說明使用基因體編輯建立突變體的流程。程序中涉及的多個步驟,需要可觀的成本和精力,例如評估 TAL 核酸酶或 CRISPR/Cas9 的活性,對帶有目標突變的個體、有相同突變的個體、兩個等位基因皆有相同突變的個體等進行分類。使用下節說明的 HMA 程序,能夠更有效率完成這些分類步驟。
確認突變:以MultiNA 系統進行異源雙股泳動試驗 (Heteroduplex Mobility Assay, HMA)
HMA
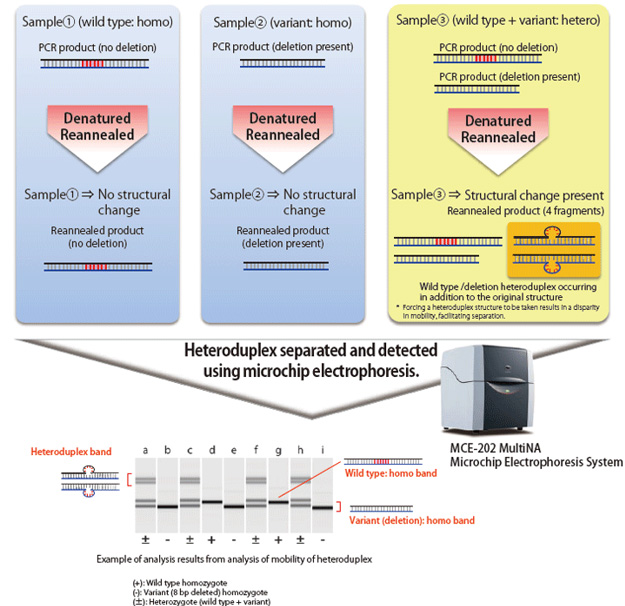
透過非同源末端接合產生突變,以達到剔除基因目的,會造成一個至幾十個鹼基的小幅度缺失或插入。要確認這類突變,需要對突變區域進行 DNA 定序;但是 DNA 定序通常費時費力,因此事前完成試樣分類更加有效率。我們利用既便利又有效之 HMA 試驗,可依據突變附近的 PCR 及電泳進行分類。
對相同長度的雙股 DNA 進行電泳時,完全互補的同源雙股 DNA 泳動情況取決於分子量。但是在異源雙股 DNA,由於部分區域發生配對錯誤,這些區域的立體結構就與同源雙股 DNA 不同,因而阻礙移行速度。HMA 程序即是利用這種現象,檢測突變是否存在。異源雙股接合的形成,是在產生突變的個體目標序列附近區域進行 PCR,以熱變性將擴增產物解離,然後使產物重新黏合。經由電泳分析,可依據電泳圖譜確定是否存在缺失或插入。
MCE-202 MultiNA DNA/RNA 分析專用 MultiNA 微晶片電泳系統
MultiNA 電泳系統可自動分析多達 108 個樣品,只需將樣品及特殊試劑裝填至系統,即可自動作業,分離效率及檢測靈敏度高於水平式電泳,而運行成本相同。MultiNA 系統進行 HMA 檢測時,能夠檢測出水平式電泳難以辨識的短插入及缺失。
實例:以異源雙股泳動試驗 (HMA) 對產生突變的個體進行分類
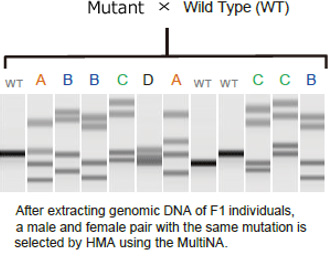
本節說明藉由 HMA 分類相同突變的 F1 雄性及雌性配對,以 MultiNA 系統獲取 F2 個體。首先從培育的 F1 個體萃取基因體 DNA,然後使用 MultiNA 系統進行 HMA,觀察到 F1 個體的多條帶分布模式,如圖所示。除了野生型品種,圖中的泳動結果另有四種模式,分別為 A、B、C、D。將這些模式的已分類雄性及雌性 F1 個體交配,可獲得 F2 個體。
由於 F2 個體定義為野生型 (wild)、異型突變體 (heterozygous mutant) 或同型突變體類型 (heterozygous mutant) (比例 1:2:1),因此可透過相同的方式,對突變體進行 HMA,並交配已分類的雄性及雌性同源突變體,以確立突變體。
詳細應用資訊Genome Editing and Creating Mutant Strains in Medaka
數據提供:京都大學農學部 Masato Kinoshita 博士



