MicrobialTrack - 特色
MALDI-TOF MS 微生物鑑定軟體
基於基因體資料建立的資料庫
MicrobialTrack 的資料庫涵蓋約 18,000 個已具有效發表分類名稱的原核生物物種(包含細菌與古菌),以及約 85,000 個依據基因組資訊所預測的原核生物物種,其中亦包含許多難以培養或尚未培養的微生物。這座龐大的資料庫大幅拓展了可鑑定的範圍,遠超越傳統 MALDI-TOF MS 微生物鑑定系統的能力。傳統上,若以核酸為基礎的技術對 96 株分離菌進行鑑定,通常需耗時約兩天;相較之下,結合 MALDI-TOF MS 與 MicrobialTrack 質譜分析軟體,可於約 3 小時內完成相同數量的鑑定,大幅提升效率與通量。
註:此處所述的處理時間係指使用甲酸與乙腈進行液態細菌培養樣品前處理的總分析時間。
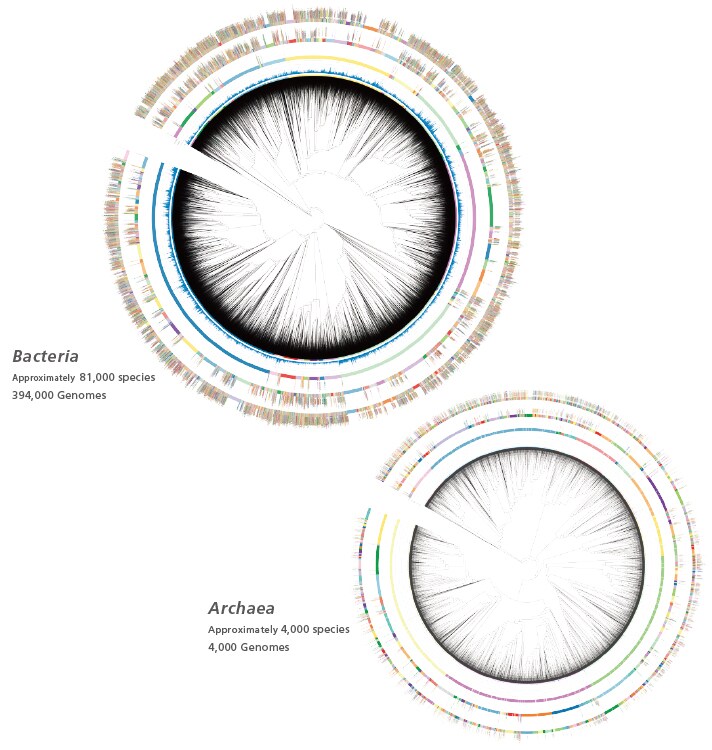
基於基因組的分類架構
MicrobialTrack 的鑑定結果會依據 Genome Taxonomy Database(GTDB, https://gtdb.ecogenomic.org) 所定義的分類名稱進行顯示。GTDB 是一套以基因組序列相似性為基礎建立的分類系統,用以界定物種層級,其主要依據兩項指標:平均核苷酸相似度(ANI, Average Nucleotide Identity):衡量兩個基因體間同源區域的序列相似程度。對齊因子(AF, Alignment Fraction):表示兩個基因體之間同源區域所佔的比例。在 GTDB 中,若兩個基因體的 ANI 高於 95%,且 AF 超過 65%,即被歸為同一物種。MicrobialTrack 採用與 GTDB 相同的標準來定義物種層級,並於其他分類階層中延續 GTDB 的分類架構與命名方式,確保鑑定結果具備高度一致性與科學依據。
MicrobialTrack 資料庫亦包含由未培養與難以培養微生物的基因體資料(例如宏基因體組裝基因組 MAGs 及單細胞組裝基因組 SAGs)所推導出的理論蛋白質質量。這些未培養微生物中,許多尚無有效發表的分類名稱,因此在 GTDB 中以暫代屬名或種名表示(例如 MGIIa-L1 或 sp002965065)。需特別注意,這些暫代名稱並非正式的分類學名稱。關於使用暫代名稱之分類群的更多資訊,可參閱 GTDB 官方網站(https://gtdb.ecogenomic.org)。隨著後續研究揭示更多基因體資訊並發表有效分類名稱,這些分類群的命名可能會有所更新。
針對不同用途的最佳化設計
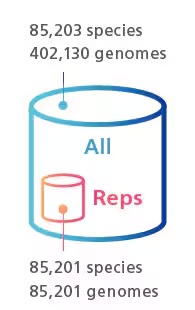
使用者可依據需求,在進行微生物鑑定時選擇兩種不同的資料庫:「All」與「Reps」。
-
「All」資料庫:收錄來自所有基因組序列所推測的理論蛋白質質量資訊。對於多數物種層級的分類群而言,「All」資料庫涵蓋來自多株菌株(即多個基因組)的蛋白質資訊。例如,Escherichia coli(大腸桿菌) 在「All」資料庫中就包含超過 30,000 筆基因組資料所推導出的蛋白質質量數據。此資料庫適用於精細搜尋與比對質譜圖,以辨識與實際樣本最為接近的菌株。
-
「Reps」資料庫:僅收錄每一物種分類群中單一具代表性的基因組所推導出的蛋白質質量資訊。此資料庫設計目的在於提供快速而概略的屬與種層級鑑定結果。透過查詢「Reps」資料庫,使用者可迅速評估量測結果與潛在候選物種之間的相似度,並獲得一份潛在比對清單,作為進一步鑑定的參考依據。
註:圖中數據點數量係根據 2025 年 3 月 的資料統計結果。
定期更新,與最新分類架構保持一致

MicrobialTrack 的資料庫會定期更新,以納入最新發現的細菌與古菌分類群,並同步採用 GTDB 最新版本的分類架構。資料庫由中央伺服器統一管理,確保使用者在授權期間內皆可無縫且即時地存取最新版本,維持鑑定準確性與時效性。
基於蛋白質體學的鑑定
MicrobialTrack 是一套利用基因組資訊推測之蛋白質理論質量資料庫,進行未知微生物鑑定的系統。其鑑定原理為:將來自微生物樣本經 MALDI-TOF MS 分析所獲得的實測質譜,與理論質量資料進行比對。隨著基因體定序技術的迅速發展,公共核酸序列資料庫1中可取得的原核生物基因體數量已大幅成長。MicrobialTrack 搭配其自主開發的大規模理論質量資料庫與獨創鑑定演算法,能在不受培養條件或 MALDI-TOF MS 儀器型號2影響的情況下,迅速且準確地鑑定多樣微生物物種。

透過雲端服務隨時隨地存取
不受地點限制,輕鬆導入
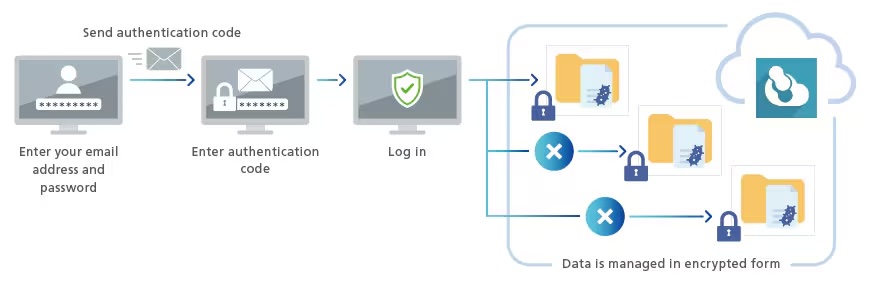
作為一項雲端服務,使用者無需購置專用電腦或安裝軟體。只要具備可連線至網際網路的環境,即可從個人電腦透過指定的 URL 進入系統並使用。
強化的安全防護
MicrobialTrack 架設於穩定且可靠的雲端基礎架構上,具備嚴謹的安全機制與存取控制。系統透過定期監控存取記錄、進行漏洞評估與軟體更新,確保資料安全,並防止使用者之間的資料互相參照,讓您能安心使用。此外,導入 多重驗證(MFA) 機制後,更可獲得更高層級的安全防護。
操作簡便
三個步驟即可完成鑑定
將微生物細胞以 MALDI-TOF MS 進行分析。由於系統支援通用格式,因此可與各廠牌儀器相容。所得的質量峰值清單會與理論質量資料庫進行比對,以鑑定微生物種類。整個鑑定流程僅需三個步驟即可完成。鑑定結果將以候選物種的排序列表呈現,並配有依鑑定信賴程度區分的彩色圖示。使用者也可檢視對應至檢測峰值的蛋白質資訊,以確認哪些蛋白質來自所鑑定的微生物。

進階的「物種複合群」鑑定
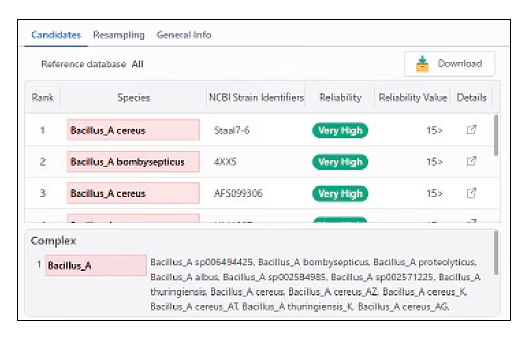
MicrobialTrack 將「物種複合群(species complex)」重新定義為由多個物種層級分類群所組成的群體,這些分類群在基因上彼此不同,但僅以理論蛋白質質量往往難以區分。MicrobialTrack 會根據理論蛋白質質量來辨識此類物種複合群,並將相關資訊通知使用者。
鑑定結果的多面向評估
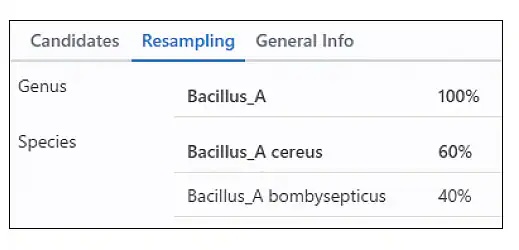
鑑定結果可能因多種因素而有所差異,包括樣品微生物的狀態、樣品的前處理方式,以及 MALDI 質譜圖的品質。因此,MicrobialTrack 採用多項性能指標,從不同角度對每一次的鑑定結果進行評估。其中一種方法是從實測峰值中隨機選取部分峰值,重複進行鑑定過程,
並顯示在重採樣過程中最常被鑑定為「最高匹配」的屬與種名稱及其出現頻率。
根據此隨機重採樣結果,軟體可對屬與種層級的鑑定可信度提供估算。
進階的蛋白質體分析平台
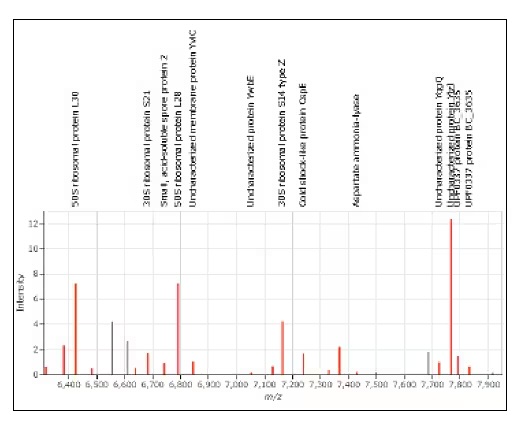
MicrobialTrack 使用資料庫中的理論質量來推測各個峰值所對應的蛋白質。推測出的蛋白質名稱會同時顯示在質譜圖上及表格式清單中。在軟體的質譜峰圖中,與理論蛋白質質量相符的質譜峰會以紅色標示,方便使用者辨識。此外,MicrobialTrack 還提供選配功能,讓使用者可檢視對應峰值的蛋白質名稱、理論質量及胺基酸序列。



